10月至今华南农业大学生命科学学院王应祥教授课题组在PNAS在线发表了题为“DNA polymerase epsilon interacts with SUVH2/9 to repress the expression of genes associated with meiotic DSB hotspot in Arabidopsis” 和“DNA Polymerase Epsilon Binds Histone H3.1-H4 and Recruits MORC1 to Mediate Meiotic Heterochromatin Condensation ”的两篇研究论文,揭示了植物减数分裂过程中DNA聚合酶epsilon(POL ε)通过和SUVH2/9互作、沉默常染色质区DNA双链断裂(DSB)位点基因的分子机制;以及POL ε特异识别组蛋白二聚体H3.1-H4并招募MORC1蛋白控制异染色质形成的分子机制。
课题组前期研究发现DNA前导链合成酶POL ε的最大亚基POL2A在减数分裂重组中具有重要功能(Huang et al., PNAS, 2015),但是其潜在的分子机制还不清楚。研究发现POL2A的N端和SUVH2/9的SET结构域互作。运用N-SIM (Structured Illumination Microscopy)观察到SUVH2在减数分裂时期与γ-H2AX标记的DSB共定位,并依赖于POL2A。通过高通量测序,发现SUVH2/9和POL2A在减数分裂细胞中共同抑制865个基因的表达,这865个基因不直接参与调控减数分裂功能,但在其转录起始位点(TSS)区域具有明显的DSB热区的特征。这暗示了POL2A和SUVH2/9可能在减数分裂DSB位置发挥基因沉默作用。该功能不像体细胞通过调控DNA的甲基化或小RNA实现,但POL2A和SUVH2/9都影响减数分裂染色体的浓缩,表明其可能直接通过调控染色质结构抑制基因表达(图1)。
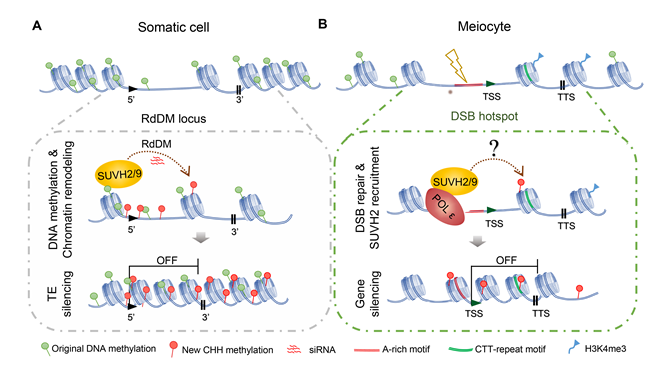
图1. POL ε-SUVH2/9调控减数分裂DSB偶联的基因沉默的模型
有趣的是,在该研究的过程中也发现了pol2a突变体影响了减数分裂中异染色质的正常浓缩。因此在第二篇文章中,研究者运用细胞学分子标记证明pol2a突变体中异染色质结构和修饰异常;通过互作筛选发现了POL2A的N端可以和浓缩蛋白ATPase MORC1相互作用,共同影响减数分裂异染色质结构和修饰,暗示了它们可能处于同一遗传通路;同时研究者还发现减数分裂时期MORC1在染色体上的定位依赖于POL2A,暗示POL2A在减数分裂时期负责招募MORC1到异染色质参与浓缩过程。进一步研究发现POL2A的C端锌指(ZF)结构域可以特异地结合组蛋白H3.1-H4的四聚体,并在不同物种中保守。通过遗传分析显示POL2A的ZF结构域对异染色质结构和H3.1的定位都至关重要;组蛋白变体H3.1上的两个特殊氨基酸A31和S87突变会影响和POL2A-ZF的互作,也会影响自身在植物体内定位。这些结果暗示了POL2A通过特异识别H3.1-H4,可能直接参与到DNA复制偶联的H3.1核小体组装中并定位到异染色质区。
综上所述,第一篇研究揭示了植物DSB位置的基因转录状态,发现了DNA聚合酶epsilon和SUVH2/9共同调控减数分裂DSB偶联的转录沉默的新机制(图1),该机制可能对于减数分裂过程的DSB修复和基因组稳定性至关重要。第二篇研究揭示了多面手DNA聚合酶POL ε参与减数分裂异染色质形成的分子机制,证明其参与DNA合成的同时,可能通过催化亚基POL2A的C端识别并参与组装组蛋白H3.1-H4形成核小体;复制完成后POL2A通过结合H3.1-H4继续停留在异染色质区并通过N端招募浓缩复合体MORC1/6,最终促进减数分裂高度浓缩的异染色质形成(图2)。

图2 POL ε调控减数分裂异染色质形成模型
岭南现代农业科学与技术广东省实验室/华南农业大学/复旦大学王应祥教授为两篇文章的通讯作者,岭南现代农业科学与技术广东省实验室/华南农业大学黄霁月教授为第一篇文章的二作以及第二篇文章的并列第一作者,复旦大学生命科学学院博士后王聪为两篇文章的第一作者。美国北卡罗来纳大学教堂山分校Gregory P. Copenhaver和美国宾夕法尼亚大学生物系马红教授为共同通讯作者,复旦大学尤辰江青年研究员为第一篇文章的共同通讯作者。复旦大学董爱武教授和中国科学院大学/中国科学院遗传与发育生物学研究所姜丹华研究员提供了指导和帮助,复旦大学研究生李颖平、张俊、何承鹏和李天杨也参与了该项工作。相关工作得到了国家自然科学基金委、复旦大学、中国博士后基金和北卡罗来纳大学的资助。
论文链接:
https://www.pnas.org/doi/10.1073/pnas.1507165112
https://www.pnas.org/doi/10.1073/pnas.2213540119
https://www.pnas.org/doi/full/10.1073/pnas.2208441119
